| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | ||||
| 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| 18 | 19 | 20 | 21 | 22 | 23 | 24 |
| 25 | 26 | 27 | 28 | 29 | 30 | 31 |
Tags
- 경사하강법
- Python강의
- 파이썬강좌
- 머신러닝공부
- java
- c언어 오목
- 딥러닝
- 파이썬강의
- 머신러닝 강의
- acmicpc.net
- 자바시작하기
- 코딩테스트
- 지도학습
- feature scaling
- 백준 알고리즘
- 비지도학습
- 선형회귀
- 인공지능
- python강좌
- 딥러닝공부
- 자바강좌
- supervised learning
- 머신러닝
- 머신러닝 강좌
- JAVA강좌
- Gradient Descent
- 효묘블로그
- 자바
- 비용함수
- unsupervised learning
Archives
- Today
- Total
컴공과컴맹효묘의블로그
k-means 알고리즘 python으로 구현해보기 (2차원 데이터 시각화) 본문
반응형
알고리즘
여기 들어가셔서 보시면 됩니다.
#주의 : 파이썬 잘 안써봐서 코드가 조금 더럽습니다. c언어 스타일로 코딩했습니다.
사용한 라이브러리
- numpy # 수학적인 데이터를 다루는 기본적인 라이브러리입니다.
- copy # deep copy를 편하게 하기위한 라이브러리입니다.
- matplotlib # 데이터를 시각화 하기 위한 라이브러리입니다.
함수들
1. n tuple 벡터에 대한 norm을 구하는 함수.
def normSquare(_x): # _x의 norm 의 제곱을 구함.
_s = 0
for _i in _x:
_s += _i ** 2
return _s
2. 전체 분산
def variance(_mu, _s): # 분산을 구함.
V = 0
for _i in range(len(_mu)):
for cor in _s[_i]:
V += normSquare(np.array(cor) - np.array(_mu[_i]))
return V
pass # V is float
3. 업데이트 된 새로운 S를 반환하는 함수
def get_reset_s(s, _mu, coord): # 업데이트 된 새로운 집합 S를 반환함.
new_s = []
for _ in range(len(s)):
new_s.append([])
for _i in coord:
_j = 0
min_mu = -1
for _d in range(len(_mu)):
dis_mu = normSquare(np.array(_i) - np.array(_mu[_d]))
if min_mu > dis_mu or min_mu == -1:
_j = _d
min_mu = dis_mu
new_s[_j].append(_i)
return new_s
pass
4. 업데이트 된 S에서 각 원소들의 무게중심으로 mu를 업데이트하는 함수. return값은 mu의 값이 변했는지의 여부
def update_mu(_mu, _s):
before_mu = copy.deepcopy(_mu)
k = 0
print(_s)
for s_i in _s:
su = np.array((0.0, 0.0))
for _i in s_i:
su += np.array(_i) / len(s_i)
# _mu[k] = copy.deepcopy(su)
for _m in range(len(_mu[k])):
_mu[k][_m] = su[_m]
k += 1
if before_mu == _mu:
return False
return True
MAIN
군집화할 k의 값과 데이터의 개수를 입력받습니다. 그리고 normal distribution을 따르는 랜덤한 값으로 x와 y를 대충 초기화 해줍니다.
k = 3
colors = ['#FF7363', '#E8935A', '#FACF71', '#EBD750', '#D1FF63']
k = int(input('input k (1<=k<=%d) : ' % (len(colors))))
if not (1 <= k <= 5):
k = 2
S = [] # k개의 집합 Si로 이루어져있음.
mu = [] # 각 클러스터의 중심점 mu k개
numData = int(input('input num of data (300 recommended) : '))
if numData <= 5:
numData = 5
half = int(numData / 2)
x = np.array((np.random.normal(-0.6, 0.3, half), np.random.normal(0.2, 0.5, half + numData % 2)))
x = np.resize(x, numData)
y = np.array((np.random.normal(-0.6, 0.3, half), np.random.normal(0.6, 0.3, half + numData % 2)))
y = np.resize(y, numData)
데이터를 튜플 형태로 합쳐주고, 각 클러스터의 중심인 \(\mu\)를 랜덤한 값으로 초기화 해주고(원래는 전체 데이터의 무게중심을 기준으로 랜덤한 값을 초기화 해줍니다. 근데 고치기 귀찮음) S에 공간을 만들어 줍니다.
data = []
for i in range(numData): # x 데이터와 y 데이터를 tuple 형태로 합침.
data.append((x[i], y[i]))
print(data)
for i in range(k): # 각 클러스트의 초기값을 랜덤으로 초기화 함.
mu.append([np.random.random(), np.random.random()])
for i in range(k): # 집합 S 공간 만듦
S.append([])
이전 S와 그 다음 S가 동일하게 될 때까지 군집화 진행.
ret = True
while ret:
S = get_reset_s(S, mu, data)
ret = update_mu(mu, S)
before그래프와 after그래프를 만들어 주고 시각화 해줍니다.
x_data = []
y_data = []
for s in S:
x_tmp = []
y_tmp = []
for si in s:
x_tmp.append(si[0])
y_tmp.append(si[1])
x_data.append(x_tmp)
y_data.append(y_tmp)
# before plot
plt.figure('before')
plt.title('BEFORE')
plt.xlim('x')
plt.ylim('y')
plt.scatter(x, y, c='#ff0000', marker='.')
plt.show()
# after plot
plt.figure('after')
plt.title('AFTER')
plt.xlim('x')
plt.ylim('y')
for n in range(len(x_data)):
plt.scatter(x_data[n], y_data[n], c=colors[n], marker='.')
print(mu)
print('Variance : ', variance(mu, S))
mu_x = [v[0] for v in mu]
mu_y = [v[1] for v in mu]
plt.plot(mu_x, mu_y, 'r*')
plt.show()
전체 코드
import numpy as np
import copy
import matplotlib.pyplot as plt
def normSquare(_x): # _x의 norm 의 제곱을 구함.
_s = 0
for _i in _x:
_s += _i ** 2
return _s
def variance(_mu, _s): # 분산을 구함.
V = 0
for _i in range(len(_mu)):
for cor in _s[_i]:
V += normSquare(np.array(cor) - np.array(_mu[_i]))
return V
pass # V is float
def get_reset_s(s, _mu, coord): # 업데이트 된 새로운 집합 S를 반환함.
new_s = []
for _ in range(len(s)):
new_s.append([])
for _i in coord:
_j = 0
min_mu = -1
for _d in range(len(_mu)):
dis_mu = normSquare(np.array(_i) - np.array(_mu[_d]))
if min_mu > dis_mu or min_mu == -1:
_j = _d
min_mu = dis_mu
new_s[_j].append(_i)
return new_s
pass
def update_mu(_mu, _s):
before_mu = copy.deepcopy(_mu)
k = 0
print(_s)
for s_i in _s:
su = np.array((0.0, 0.0))
for _i in s_i:
su += np.array(_i) / len(s_i)
# _mu[k] = copy.deepcopy(su)
for _m in range(len(_mu[k])):
_mu[k][_m] = su[_m]
k += 1
if before_mu == _mu:
return False
return True
############################################## MAIN
k = 3
colors = ['#FF7363', '#E8935A', '#FACF71', '#EBD750', '#D1FF63']
k = int(input('input k (1<=k<=%d) : ' % (len(colors))))
if not (1 <= k <= 5):
k = 2
S = [] # k개의 집합 Si로 이루어져있음.
mu = [] # 각 클러스터의 중심점 mu k개
numData = int(input('input num of data (300 recommended) : '))
if numData <= 5:
numData = 5
half = int(numData / 2)
x = np.array((np.random.normal(-0.6, 0.3, half), np.random.normal(0.2, 0.5, half + numData % 2)))
x = np.resize(x, numData)
y = np.array((np.random.normal(-0.6, 0.3, half), np.random.normal(0.6, 0.3, half + numData % 2)))
y = np.resize(y, numData)
data = []
for i in range(numData): # x 데이터와 y 데이터를 tuple 형태로 합침.
data.append((x[i], y[i]))
print(data)
for i in range(k): # 각 클러스트의 초기값을 랜덤으로 초기화 함.
mu.append([np.random.random(), np.random.random()])
for i in range(k): # 집합 S 공간 만듦
S.append([])
ret = True
while ret:
S = get_reset_s(S, mu, data)
ret = update_mu(mu, S)
x_data = []
y_data = []
for s in S:
x_tmp = []
y_tmp = []
for si in s:
x_tmp.append(si[0])
y_tmp.append(si[1])
x_data.append(x_tmp)
y_data.append(y_tmp)
# before plot
plt.figure('before')
plt.title('BEFORE')
plt.xlabel('x')
plt.ylabel('y')
plt.scatter(x, y, c='#ff0000', marker='.')
plt.show()
# after plot
plt.figure('after')
plt.title('AFTER')
plt.xlabel('x')
plt.ylabel('y')
for n in range(len(x_data)):
plt.scatter(x_data[n], y_data[n], c=colors[n], marker='.')
print(mu)
print('Variance : ', variance(mu, S))
mu_x = [v[0] for v in mu]
mu_y = [v[1] for v in mu]
plt.plot(mu_x, mu_y, 'r*')
plt.show()
결과창
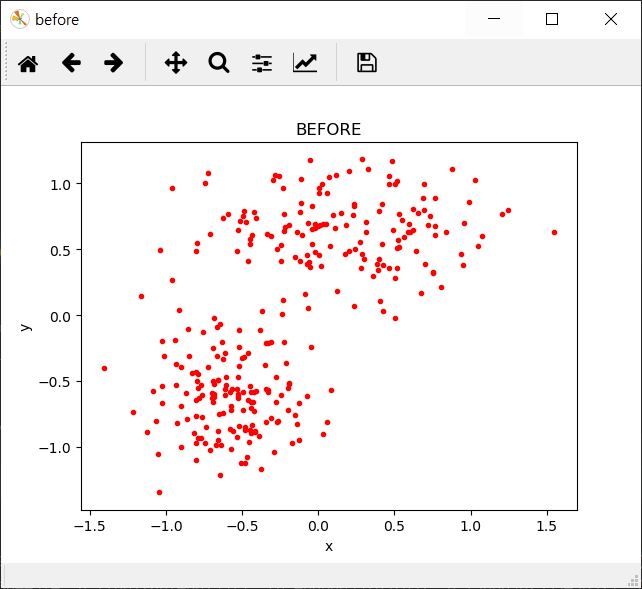
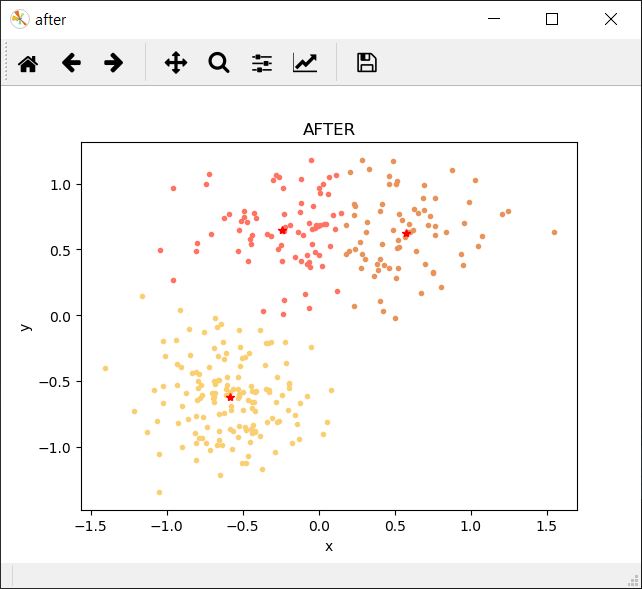
k-means algorithm의 한계점...
- 클러스터 개수 k의 값을 입력 파라미터로 지정해주어야 한다. 예를 들어 위 사진은 실제로(직관적으로도) 클러스터의 수는 2개인데, k의 값을 3으로 입력한 경우이다. k의 값에 따라 결과가 극명하게 달라지며, 때로는 좋지 못한 결과를 보여줄 가능성이 있다.
- global minimum이 아닌 local minimun으로 수렴할 수 있다. 초기 \(\mu\)의 값이 잘못 설정되면 알고리즘의 최적값이 local minimum으로 수렴할 수 있다. 이 경우 담금질 기법이나 \(\mu\)의 초기값을 다르게해서 여러번 시도하는 방법으로 이 문제를 완화할 수 있다.
사진 출처 위키피디아
- 이상값(outlier)에 민감하다. k-mean 알고리즘은 각 집합의 원소들의 무게중심을 기준으로 클러스터링을 하는데, 이 때 멀리 떨어져있는 데이터 때문에 무게중심이 클러스터가 이상값 쪽으로 치우질 수 있다. 해결 방법으로는 사전에 이상값을 제거하거나, k-medoids algorithm을 이용하면 이상값의 영향을 줄일 수 있다.
- 구형(spherical)이 아닌 클러스터를 찾는 데에는 적합하지 않다. k-means 알고리즘은 비용함수를 계산할 때 중심점과 각 데이터 오브젝트간의 거리를 계산한다. 이때 사용되는 거리는 유클리드 거리이다. 따라서, 알고리즘 수행 시 중심점으로부터 구형으로 군집화가 이루어진다. 만약 주어진 데이터 집합의 분포가 구형이 아니라면 클러스터링 결과가 예상과 다를 수 있다.
사진 출처 위키피디아
반응형
'컴퓨터 > 머신러닝' 카테고리의 다른 글
| DBSCAN을 이용한 식당 리뷰 요약 서비스 (6) | 2025.06.08 |
|---|---|
| 수알못의 머신러닝 공부 : Logistic Regression을 이용한 Classification을 Python으로 구현 (0) | 2020.04.02 |
| 수알못의 머신러닝 : 선형회귀 - normal equation 정규방정식 (0) | 2019.08.23 |
| Polynomail Regression 다항식 회귀 (0) | 2019.08.19 |
| 머신러닝 회귀 분석 Feature Scaling (0) | 2019.08.19 |
Comments






